10 “理想”scRANA-seq流程
10.1 实验设计
- 避免混淆生物效应和批次效应(Figure 10.1)
- 如果可以,同一实验多个条件
- 每个条件多次重复,如果可以,不同条件重复一起执行
- 统计不能完全校正完全混乱的实验!
- 唯一分子标识符(UMI)
- 大大降低数据中的噪音
- 可能会降低基因检测率
- 丢失剪切信息
- 使用更长的UMIs(~10bp)
- 使用UMI-tools校正测序误差
- Spike-ins
- 有助于质量控制
- 可能对标准化read counts很有用
- 可用于近似细胞大小/ RNA含量(如果与生物学问题相关)
- 通常表现出比内源基因更高的噪音(移液错误,混合物质量)
- 需要更深的测序才能获得每个细胞足够的内源性reads
- 细胞数量 vs Read深度
- 基因检测平台每个细胞1百万reads数
- 检测转录因子(调节网络)需要测序深度神和最敏感的protocol(比如Fluidigm C1)
- 细胞聚类和细胞类型鉴定受益于大量细胞,并且不需要高测序深度(每个细胞约100,000个reads)
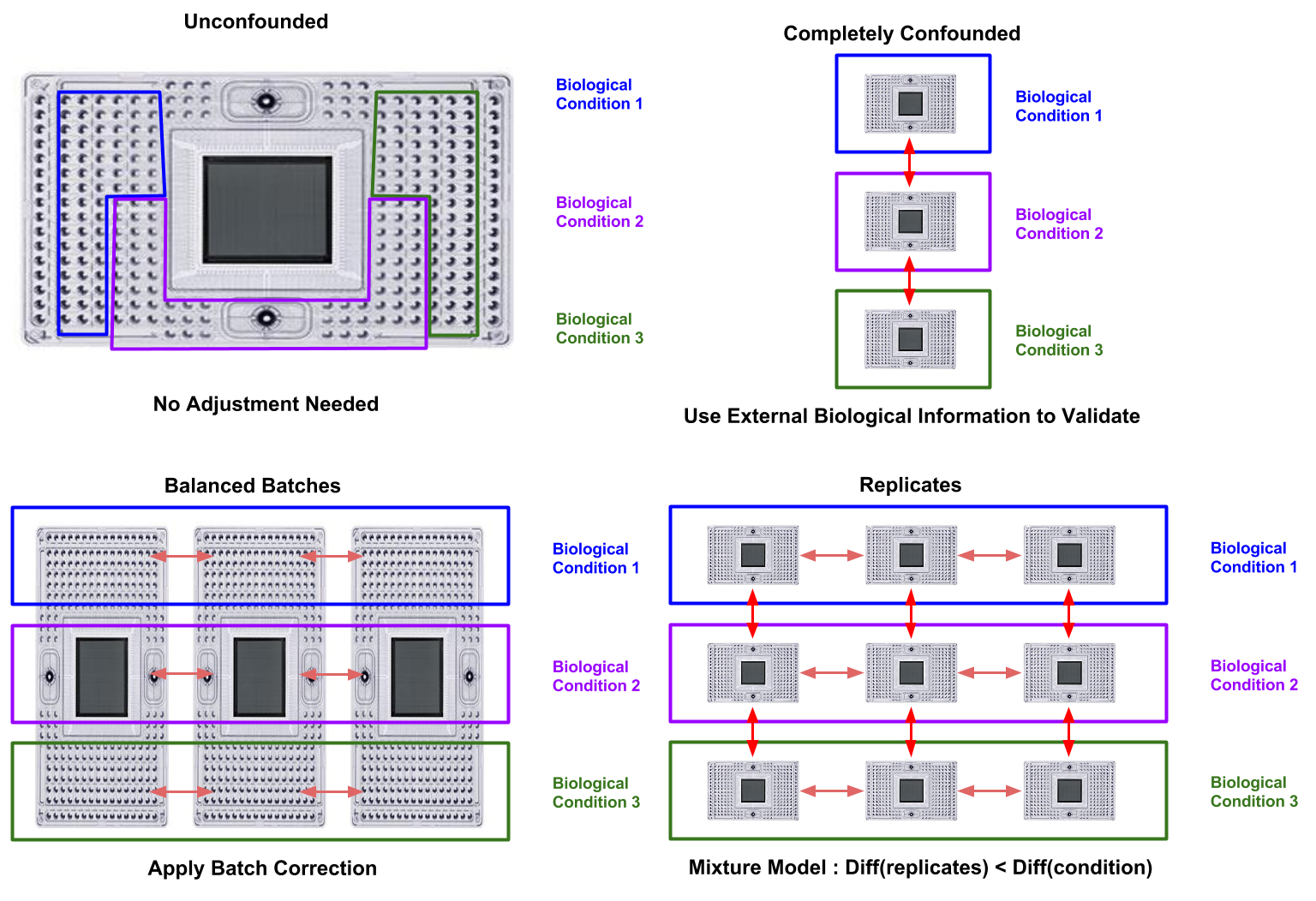
Figure 10.1: Appropriate approaches to batch effects in scRNASeq. Red arrows indicate batch effects which are (pale) or are not (vibrant) correctable through batch-correction.
10.2 reads处理
- 质控 & Trimming
- 比对
- 定量
- 小数据集,不是UMIs : featureCounts
- 大数据集,不是UMIs: Salmon, kallisto
- UMI数据集: UMI-tools + featureCounts
10.3 准备表达矩阵
- 细胞质控
- scater
- 考虑:mtRNA,rRNA,spike-ins(如果有的话),每个细胞检测到的基因数,每个细胞的总reads/分子数
- 文库大小标准化
- 校正批次效应
- 重复/混淆因子 RUVs
- 未知或不平衡的生物群 mnnCorrect
- 平衡设计 ComBat