2 scRNA-seq介绍
2.1 Bulk RNA-seq
- 2000后期重大突破,取代微阵列芯片被广泛采用
- 测量大量混合细胞的平均表达水平
- 用于比较转录组学,例如比较不同物种同一组织的样本
- 用于量化整体表达特征,例如疾病研究
- 不足研究异质性系统,例如早起发育的研究,复杂组织(脑)的研究
- 不能呈现基因表达随机性
2.2 scRNA-seq
- 一项新兴技术,第一篇文章为汤富酬发表在Nature method (Tang et al. 2009)
- 直到~2014年,新测序手段和更低的成本使其流行起来
- 在大量细胞检测每个基因的表达水平
- 可以研究新的生物学问题,其中转录组的细胞特异性变化是重要的,比如细胞类型鉴定,细胞响应的异质性,基因表达的随机性,细胞间基因调控网络的推断
- 研究细胞数目从\(10^2\)到\(10^6\)个细胞,而且每年递增
- 目前有不同单细胞的protocol,比如SMART-seq2 (Picelli et al. 2013), CELL-seq (Hashimshony et al. 2012) 和 Drop-seq (Macosko et al. 2015)
- 也有商业平台, 包括 Fluidigm C1, Wafergen ICELL8 和 10X Genomics Chromium
- 一些计算分析bulk RNA-seq的方法也适用于 scRNA-seq
- 大多数情况下 scRNA-seq计算分析需要调整已有方法或者开发新方法
2.3 工作流程
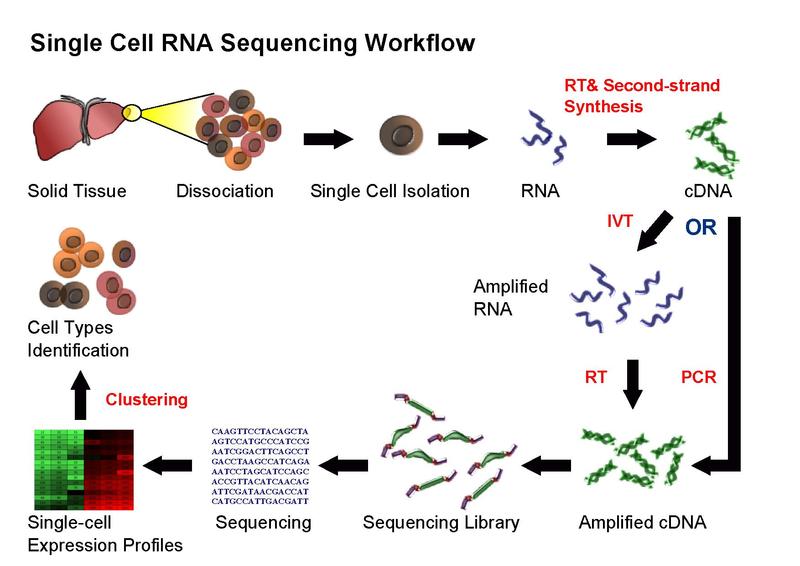
Figure 2.1: 单细胞测序流程 (源自wiki)
总体而言,scRNA-seq实验protocol和bulk RNA-seq类似,我们将在下一章讨论一些最常用的方法。
2.4 计算分析
本课程是用scRNA-seq实验获得的数据进行计算分析。
- 第一步(黄色)对任何高通量测序数据都是通用的;
- 后续步骤(橙色)需要整合现有的RNA-Seq分析方法和新方法来解决scRNA-Seq的技术差异;
- 最后步骤(蓝色),用专门为scRNA-Seq开发的方法进行生物学解释。
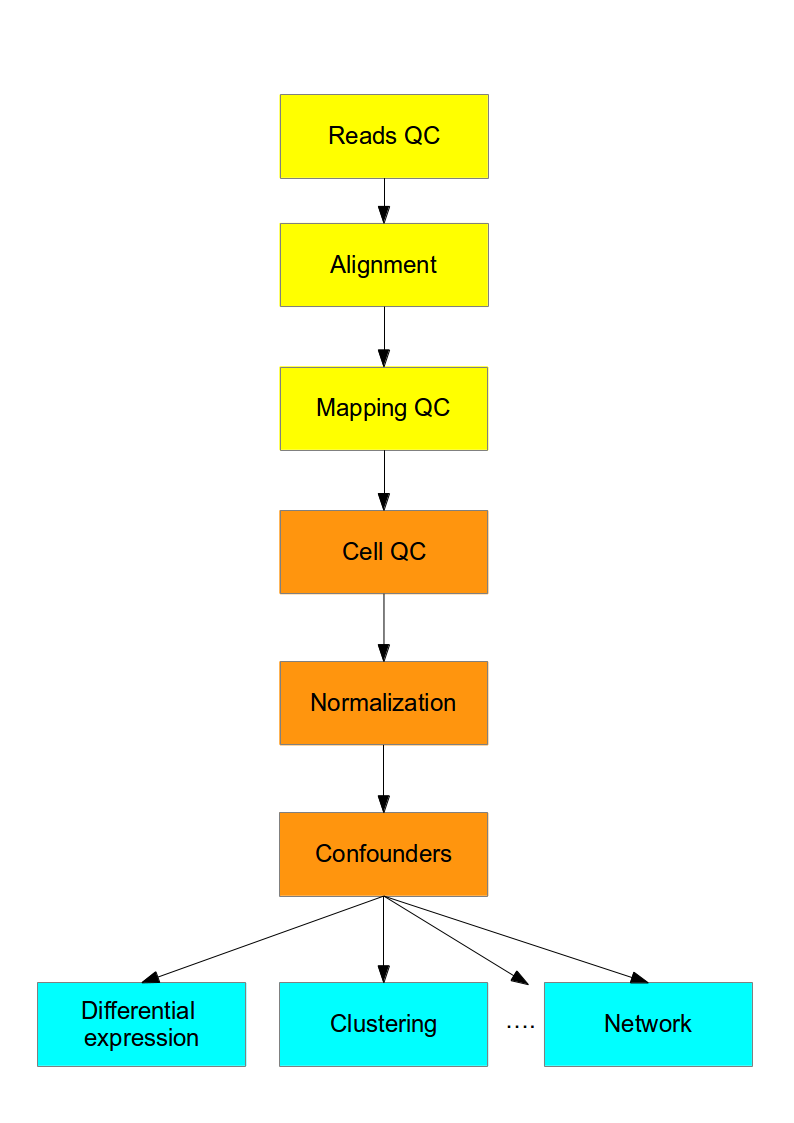
Figure 2.2: scRNA-seq分析流程图
目前有几篇关于scRNA-seq分析的综述文章,包括 (Stegle, Teichmann, and Marioni 2015)
目前,也有不同的平台执行上述流程图的一步或多个步骤,包括:
- Falco 云端单细胞RNA-seq处理框架
- SCONE (Single-Cell Overview of Normalized Expression), 单细胞RNA-seq数据质量控制和标准化的包.
- Seurat 用于单细胞RNA-seq数据质量控制,分析和数据探索的R包.
- ASAP (Automated Single-cell Analysis Pipeline) 是一个单细胞分析交互式webserver.
- Bioconductor 是一个分析高通量基因组数据开源,开放式软件项目,包括分析单细胞数据的工具包。
2.5 挑战
scRNA-seq和bulk RNA-seq最大的不同在于每个测序文库是单个细胞,而不是一群细胞。因此需要各位注意来自不同细胞(测序文库)的结果的比较。文库之间差异的主要来源是:
- 扩增偏好性和扩增效率 (高达1,000,000倍)
- 基因的’dropout’,其中一个基因在一个细胞中等表达水平,但在另外一个细胞没有检测到(Kharchenko, Silberstein, and Scadden 2014).
在两种情况下, 由于RNA仅来自一个细胞,因此低起始量的转录本是导致差异的主要原因。提高转录本捕获效率和减少扩增偏差是目前活跃的研究灵越。从后续的课程我们也可以看到,可以通过适当的标准化和校正方法来缓解这些问题。
2.6 实验方法
)](figures/moores-law.png)
Figure 2.3: 单细胞转录组学摩尔定律 (图片来自 Svensson et al)
开发新的scRNA-seq方法和protocol是目前非常活跃的一个研究领域,在过去的几年中已经发表了一些protocol,不完全列表如下:
- CEL-seq (Hashimshony et al. 2012)
- CEL-seq2 (Hashimshony et al. 2016)
- Drop-seq (Macosko et al. 2015)
- InDrop-seq (Klein et al. 2015)
- MARS-seq (Jaitin et al. 2014)
- SCRB-seq (Soumillon et al. 2014)
- Seq-well (Gierahn et al. 2017)
- Smart-seq (Picelli et al. 2014)
- Smart-seq2 (Picelli et al. 2014)
- SMARTer
- STRT-seq (Islam et al. 2013)
这些方法可以按照不同方法进行归类,最重要的两个方面是定量和捕获
定量有两种类型,全长和基于标签。前者试图达到每个转录本均匀覆盖率;然而基于tag的protocol只捕获每个RNA的5’或3’端。量化方法的选择对于数据可用于何种类型的分析具有重要意义。理论上讲,基于全长的protocol可以对整个转录本进行均匀测序,然而通常测序覆盖有偏差。基于tag的protocol主要优势是其可以和唯一的分子标识符结合使用,提高定量准确性(查看章节 4.6)。另一方面,测序限制在转录组的一端,降低比对map率,并难以区分不同的异构体(Archer et al. 2016)。
捕获的策略决定了通量,细胞如何被选择以及除测序外还可以获得哪种附加信息。三种常用的方法是基于微孔(microwell)-,微流体(microfluidic)-,液滴(droplet)-的方法。

Figure 2.4: 微孔板 (图片来自wiki)
对于基于微孔的平台,使用例如移液管或激光捕获分离细胞并置于微流体孔中。 基于微孔的方法的一个优点是它们可以与荧光激活细胞分选(FACS)结合,基于表面标记物分选细胞。 因此,当想要分离特定细胞群体用于测序时,该策略非常有用。 另一个优点是可以拍摄细胞的照片。 该图像提供了额外的细胞形态,适用于识别包含受损细胞或双份细胞的微孔。 这些方法的主要缺点是它们通常是通量低并且每个单细胞所需的工作量可能相当大。

Figure 2.5: 96孔Fluidigm C1芯片 (图片来自Fluidigm)
微流体平台, 比如 Fluidigm’s C1, 提供了更加集成的系统,用于捕获细胞和进行文库制备所需的过程。 因此,它们提供比基于微孔的平台更高的通量。 通常,在微流体平台中仅捕获约10%的细胞,因此不适合处理稀有细胞类型或非常少量的细胞。 此外,芯片相对昂贵,但由于反应可以以较小的体积进行,因此可以节省试剂。
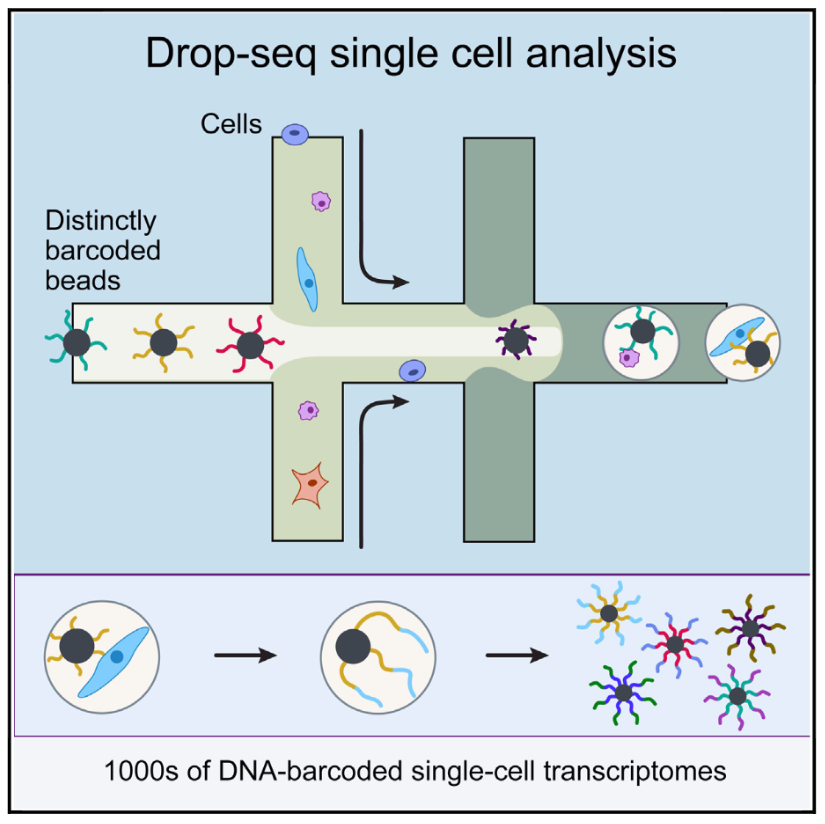
Figure 2.6: drop-seq方法的原理图 (图片来自Macosko et al)
基于液滴的方法将每个单独的细胞与包括建库所需酶的珠子(bead)一起封装在纳升液滴内。每个珠子都包含唯一的条形码(barcode),加到所有来自该细胞的序列上。因此可以合并所有液滴进行测序,再基于barcode将序列分配给不同的细胞。Droplet平台通常具有最高的通量,因为建库准备成本约为\(.05\) 美元/细胞。相反,测序成本成为其限制因素,通常实验覆盖率低,仅检测到几千个转录本 (Ziegenhain et al. 2017)。
2.7 如何选择合适的平台
最合适的平台取决于手头的生物学问题。 例如,如果研究组织的组成,那么将允许捕获非常大量细胞的基于液滴的方法可能是最合适的。 另一方面,如果人们对具有已知表面标记物的稀有细胞类群感兴趣,那么最好使用FACS进行富集,然后对较少数量的细胞进行测序。
显然,如果研究不同的异构体,那么全长转录物定量将更合适。相比之下,UMI只能与基于tag的protocol一起使用,促进基因水平的量化。
来自Enard group (Ziegenhain et al. 2017)和Teichmann group (Svensson et al. 2017)最近两项研究比较了几种不同的protocol。Ziegenhain等人在同一小鼠胚胎干细胞样本(mESCs)上比较了五种不同的protocol。通过控制细胞数量和测序深度,作者能够直接比较不同protocol的灵敏度,噪声水平和成本。 他们的结论的一个例子如下图所示,不同protocol检测的基因数量(对于给定的检测阈值)差别很大。drop-seq和Smart-seq2之间几乎有两倍的差异,这表明protocol的选择会对研究有重大影响。
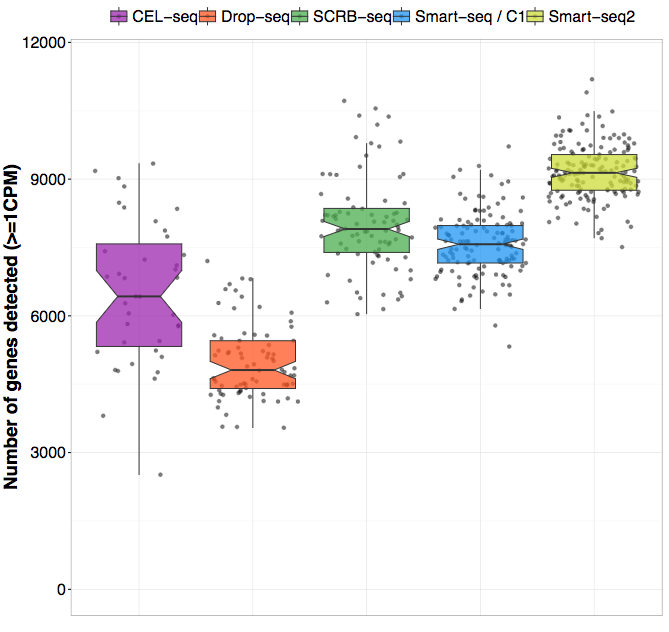
Figure 2.7: Enard group 研究
Svensson等采用了另外一种方法,通过使用已知浓度的合成转录本(spike-ins,后面详细介绍)来测量不同protocol的准确性和灵敏度。通过广泛的比较研究,他们同样发现不同protocol间区别较大。
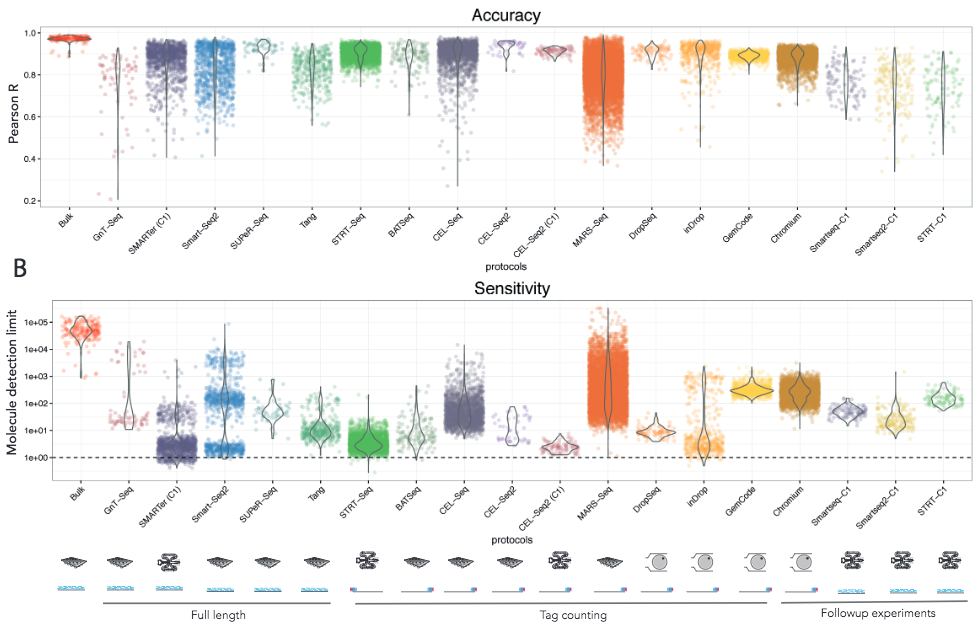
Figure 2.8: Teichmann group 研究
随着实验技术的发展和用于量化技术噪声的计算方法的改进,后续研究有助于我们进一步了解不同方法的优缺点。因为基准测试可以确定哪些策略是最有用的,这些比较研究不仅有助于研究人员决定使用哪种protocol,而且有助于开发新方法。
References
Archer, Nathan, Mark D. Walsh, Vahid Shahrezaei, and Daniel Hebenstreit. 2016. “Modeling Enzyme Processivity Reveals That RNA-Seq Libraries Are Biased in Characteristic and Correctable Ways.” Cell Systems 3 (5): 467–479.e12. https://doi.org/10.1016/j.cels.2016.10.012.
Gierahn, Todd M, Marc H Wadsworth 2nd, Travis K Hughes, Bryan D Bryson, Andrew Butler, Rahul Satija, Sarah Fortune, J Christopher Love, and Alex K Shalek. 2017. “Seq-Well: Portable, Low-Cost RNA Sequencing of Single Cells at High Throughput.” Nat. Methods 14 (4): 395–98.
Hashimshony, Tamar, Naftalie Senderovich, Gal Avital, Agnes Klochendler, Yaron de Leeuw, Leon Anavy, Dave Gennert, et al. 2016. “CEL-Seq2: Sensitive Highly-Multiplexed Single-Cell RNA-Seq.” Genome Biol 17 (1). https://doi.org/10.1186/s13059-016-0938-8.
Hashimshony, Tamar, Florian Wagner, Noa Sher, and Itai Yanai. 2012. “CEL-Seq: Single-cell RNA-Seq by Multiplexed Linear Amplification.” Cell Reports 2 (3): 666–73. https://doi.org/10.1016/j.celrep.2012.08.003.
Islam, Saiful, Amit Zeisel, Simon Joost, Gioele La Manno, Pawel Zajac, Maria Kasper, Peter Lönnerberg, and Sten Linnarsson. 2013. “Quantitative Single-Cell RNA-Seq with Unique Molecular Identifiers.” Nat Meth 11 (2): 163–66. https://doi.org/10.1038/nmeth.2772.
Jaitin, Diego Adhemar, Ephraim Kenigsberg, Hadas Keren-Shaul, Naama Elefant, Franziska Paul, Irina Zaretsky, Alexander Mildner, et al. 2014. “Massively Parallel Single-Cell RNA-seq for Marker-Free Decomposition of Tissues into Cell Types.” Science 343 (6172): 776–79.
Kharchenko, Peter V, Lev Silberstein, and David T Scadden. 2014. “Bayesian Approach to Single-Cell Differential Expression Analysis.” Nat Meth 11 (7): 740–42. https://doi.org/10.1038/nmeth.2967.
Klein, Allon M., Linas Mazutis, Ilke Akartuna, Naren Tallapragada, Adrian Veres, Victor Li, Leonid Peshkin, David A. Weitz, and Marc W. Kirschner. 2015. “Droplet Barcoding for Single-Cell Transcriptomics Applied to Embryonic Stem Cells.” Cell 161 (5): 1187–1201. https://doi.org/10.1016/j.cell.2015.04.044.
Macosko, Evan Z., Anindita Basu, Rahul Satija, James Nemesh, Karthik Shekhar, Melissa Goldman, Itay Tirosh, et al. 2015. “Highly Parallel Genome-Wide Expression Profiling of Individual Cells Using Nanoliter Droplets.” Cell 161 (5): 1202–14. https://doi.org/10.1016/j.cell.2015.05.002.
Picelli, Simone, Åsa K Björklund, Omid R Faridani, Sven Sagasser, Gösta Winberg, and Rickard Sandberg. 2013. “Smart-Seq2 for Sensitive Full-Length Transcriptome Profiling in Single Cells.” Nat Meth 10 (11): 1096–8. https://doi.org/10.1038/nmeth.2639.
Picelli, Simone, Omid R Faridani, Asa K Björklund, Gösta Winberg, Sven Sagasser, and Rickard Sandberg. 2014. “Full-Length RNA-seq from Single Cells Using Smart-Seq2.” Nat. Protoc. 9 (1): 171–81.
Soumillon, Magali, Davide Cacchiarelli, Stefan Semrau, Alexander van Oudenaarden, and Tarjei S Mikkelsen. 2014. “Characterization of Directed Differentiation by High-Throughput Single-Cell RNA-Seq.” bioRxiv, March, 003236.
Stegle, Oliver, Sarah A. Teichmann, and John C. Marioni. 2015. “Computational and Analytical Challenges in Single-Cell Transcriptomics.” Nat Rev Genet 16 (3): 133–45. https://doi.org/10.1038/nrg3833.
Svensson, Valentine, Kedar Nath Natarajan, Lam-Ha Ly, Ricardo J Miragaia, Charlotte Labalette, Iain C Macaulay, Ana Cvejic, and Sarah A Teichmann. 2017. “Power Analysis of Single-Cell RNA-Sequencing Experiments.” Nat Meth 14 (4): 381–87. https://doi.org/10.1038/nmeth.4220.
Tang, Fuchou, Catalin Barbacioru, Yangzhou Wang, Ellen Nordman, Clarence Lee, Nanlan Xu, Xiaohui Wang, et al. 2009. “mRNA-Seq Whole-Transcriptome Analysis of a Single Cell.” Nat Meth 6 (5): 377–82. https://doi.org/10.1038/nmeth.1315.
Ziegenhain, Christoph, Beate Vieth, Swati Parekh, Björn Reinius, Amy Guillaumet-Adkins, Martha Smets, Heinrich Leonhardt, Holger Heyn, Ines Hellmann, and Wolfgang Enard. 2017. “Comparative Analysis of Single-Cell RNA Sequencing Methods.” Molecular Cell 65 (4): 631–643.e4. https://doi.org/10.1016/j.molcel.2017.01.023.